SynBio Column Recent Research Roundup | RRR [EP.1]
พิมพ์หนังสือทั้งเล่มด้วยดีเอ็นเอ
จดหมายพัสดุของคุณกำลังถูกส่งไปที่บ้านของคุณภายในเดือนกุมภาพันธ์ปีนี้ นี่เป็นข้อความที่เข้ามาในอีเมลของผมหลังจากสั่งซื้อหนังสือที่เขียนลงบนดีเอ็นเอ (DNA) เล่มแรกของ Asimov Press(1) สำนักข่าวออนไลน์ที่ตั้งอยู่ในเมืองบอสตัน สหรัฐอเมริกา ที่ผลิตผลงานทางด้านชีววิทยาสังเคราะห์ ปัญญาประดิษฐ์ และสาขาที่เกี่ยวข้องออกมามากมายในช่วงหลายปีที่ผ่านมา ในวันที่เขียนบทความนี้ ตัวผมที่ทำงานและอาศัยอยู่ในโซนบอสตันสามารถสั่งซื้อหนังสือทั้งในรูปแบบกระดาษและ DNA ได้ในราคา $60 รวมกับค่าส่งอีก $5 โดยที่ตัวหนังสือ DNA นี้เริ่มเดินทางจากบอสตันในการพิมพ์หนังสือลงบนชิ้นส่วน DNA โดยบริษัท CATALOG ที่เชี่ยวชาญเรื่อง DNA computing แล้วส่งหนังสือในรูป DNA ไปที่ฝรั่งเศสเพื่อให้บริษัท Imagene บรรจุลงในหลอดแคปซูลโลหะด้วยเทคโนโลยี DNAshell ที่เพิ่มความเสถียรของตัว DNA เหล่านี้ จากนั้นจึงส่งหลอดแคปซูลส่วนหนึ่งกลับไปที่แคลิฟอร์เนียเพื่อให้บริษัท Plasmidsaurus อ่านหนังสือ DNA ชุดนี้ด้วยเทคโนโลยี Oxford Nanopore Technologies (ONT) sequencing การกดซื้อหนังสือ DNA เล่มนี้ชวนให้ผมอ่านเรื่องราวอื่น ๆ เกี่ยวกับเทคโนโลยีการเก็บข้อมูลในรูปแบบของ DNA ขึ้นมาหลาย order of magnitude ดังนั้นผมจึงอยากจะหยิบเอาผลงานวิชาการที่เพิ่งออกมาในปี 2024 มาแชร์ให้ทุกคนตื่นเต้นไปด้วยกันครับ
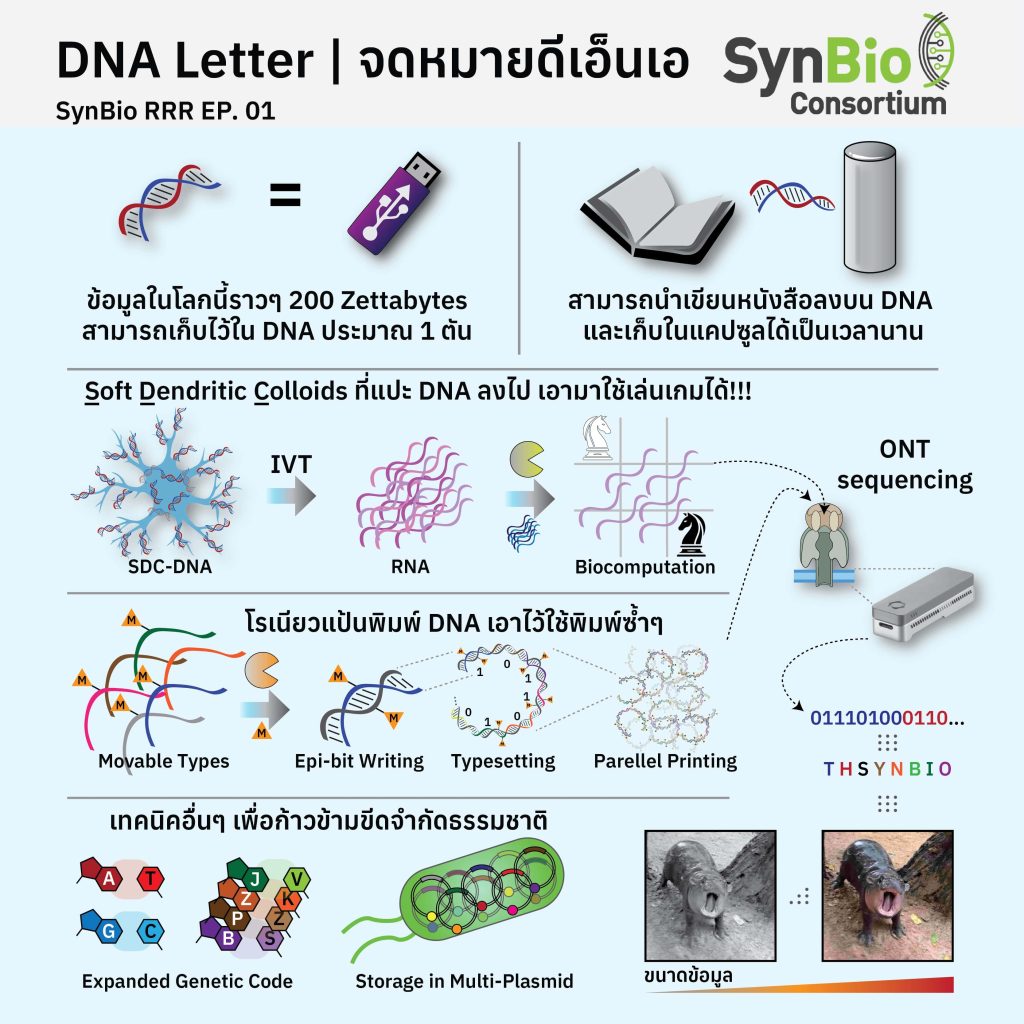
ในปี 2025 โลกเราจะมีข้อมูลมากถึง 200 โกฏิปโกฏิไบต์
ความพิเศษของลำดับ DNA ในมุมมองของการเก็บข้อมูลคือการที่ธรรมชาติใช้รหัสอยู่ 4 แบบที่มีโครงสร้างทางเคมีต่างกัน (A, T, G, และ C) ทำให้เราสามารถเก็บข้อมูลต่อหนึ่งหน่วยนับได้ซับซ้อนกว่าเดิมเมื่อเทียบกับการเก็บข้อมูลเป็น bits (0, 1) ที่มาจากรหัสเปิด-ปิดในวงจรคอมพิวเตอร์ ในยุคที่ Generative AI ส่งผลต่อการเกิดใหม่ของข้อมูลแบบก้าวกระโดด การพัฒนาระบบจัดเก็บข้อมูลจึงเป็นอีกประเด็นสำคัญในวงการวิทยาศาสตร์และเทคโนโลยี อ้างอิงจาก Cybersecurity Ventures(2) มีการคาดการว่าในปี 2025 นี้จะมีปริมาณข้อมูลมากถึง 200 Zettabytes ที่โลกของเราต้องหาที่จัดเก็บ หน่วย Zetta นี้คือหลัก 10 ยกกำลัง 21 นับแบบคำไทยก็มากถึงโกฏิปโกฏิไบต์ (จำยากกว่า Zetta อีก แต่ยังไม่มากโข เพราะมากโขน่าจะเพี้ยนมาจากอักโขภิณีที่นับด้วย 10 ยกกำลัง 42 จะเยอะไปไหน!!!) ซึ่งให้จินตนาการออกมาก็คงยาก เอาเป็นว่าประมาณเก็บเพลง mp3 ได้ราว ๆ เจ็ดล้านล้านเพลง ความบันเทิงของการเก็บข้อมูลใน DNA นี้ก็คือเราอาจจะให้ห้องแค่ห้องเดียวในการเก็บข้อมูลกว่าสองร้อยโกฏิปโกฏิไบต์นี้ ผมเอาไปถาม AI ดูแล้วมันบอกมาว่าน่าจะใช้ DNA ประมาณเกือบ ๆ หนึ่งตัน (1000 กิโลกรัม) สำหรับเทคโนโลยีการเก็บ เขียน และอ่าน DNA ในปัจจุบัน
เราลองมาชวนคุยถึงพัฒนาการของระบบการเก็บ เขียน และอ่าน DNA กันอีกหน่อย การเก็บ DNA นี้หากมีข้อได้เปรียบจากธรรมชาติอยู่ก่อนแล้ว เพราะความเสถียรของ DNA นี้มากพอให้เราสกัดออกมาจากซากอารยธรรมโบราณหลักล้านปีก่อนเพื่อนำมาศึกษาได้ หาก DNA ถูกเก็บในสภาพแวดล้อมที่เหมาะสม คืออุณหภูมิและความชื้นต่ำ มันอาจจะคงอยู่ได้นับล้านปี เทคโนโลยีการอ่าน DNA ก็มีการพัฒนาแบบก้าวกระโดดนับตั้งแต่การอ่านด้วยวิธิ Sanger sequencing และการพัฒนา Next-Generation sequencing ที่ผลักดันโครงการ Human Genome Project ให้เสร็จก่อนเวลาที่คาดการณ์ไว้ได้ จนกระทั่งเทคโนโลยียุคล่าสุดอย่าง Nanopore sequencing ที่ใช้ในการอ่านหนังสือ DNA ข้างต้น แต่ทว่าการเขียนด้วย DNA นี้ยังตามหลังเทคโนโลยีอื่น ๆ อยู่มากทั้งในด้านความเร็ว ความยาวของสาย DNA ไปจนถึงราคาที่ต้องจ่ายต่อตัวอักษร DNA ที่พิมพ์ออกมา อย่างไรก็ตามเทคโนโลยีเหล่านี้มีการอัพเกรดขึ้นมาเรื่อย ๆ ในยุคที่เทคโนโลยีชีววิทยาสังเคราะห์เริ่มออกสู่ตลาดและสร้างแรงผลักดันให้กับวงการในช่วงยี่สิบปีที่ผ่านมา ดังนั้นงานในด้าน DNA Data Storage จึงมักเน้นไปที่กระบวนการ (algorithm) ในการเขียน อ่าน และจัดเก็บชุดข้อมูลให้มีศักยภาพและง่ายต่อการนำมาใช้มายิ่งขึ้น บทความนี้จะหยิบงานที่เพิ่งตีพิมพ์ในด้านนี้มาเล่าให้ฟังแบบคร่าว ๆ
แก้โจทย์หมากรุกด้วยการแปะ DNA ไว้บนวัสดุโครงตาข่าย
ในช่วงกลางปี 2024 ทีมวิจัยจาก North Carolina State University และ Johns Hopkins University ได้ตีพิมพ์ผลงานเกี่ยวกับการจัดเก็บ อ่าน และประมวลผลด้วย DNA โดยผสมผสานเข้ากับเทคโนโลยีวัสดุ Soft Dendritic Colloids (SDCs)(3) ที่มีโครงร่างตาข่ายคล้ายกับเซลล์ประสาท มาช่วยจับ DNA เพื่อเพิ่มเสถียรภาพในการจัดเก็บทั้งในรูปแบบของแข็งและสารละลาย ทั้งยังเพิ่มความสะดวกในการอ่าน ลบ และเขียนใหม่ การอ่านข้อมูล DNA ถูกยึดอยู่กับ SDC นี้สามารถเข้าถึงได้ด้วยการ transcribe นอกเซลล์ หรือ in vitro transcription (IVT) ให้ DNA กลายเป็น RNA คล้ายกับการอ่านข้อมูล DNA ของสิ่งมีชีวิตเอง ซึ่งกระบวนการนี้สามารถแยก RNA ออกจาก DNA ได้ง่าย ๆ ด้วยโมเลกุลแม่เหล็กที่ช่วยยึดเอาวัสดุ SDC-DNA นี้เอาไว้ในช่องเล็ก ๆ (microfluidic) และนำเอา RNA ที่ว่านี้มาคัดลอกเป็น DNA เพื่ออ่านข้อมูลด้วยวิธี Next-Generation sequencing หรือนำ RNA ไปอ่านด้วยเทคโนโลยี Nanopore โดยตรงเลยก็ได้ เทคโนโลยีการอ่าน RNA sequence ด้วย ONT นี้เป็นระบบที่ค่อนข้างใหม่ ทางทีมวิจัยจึงต้องร่วมพัฒนาระบบการทำงานในส่วนนี้เช่นกัน นอกจากนี้หากต้องการลบดีเอ็นเอทั้งหมด หรือบางส่วน ออกจาก SDC-DNA ก็สามารถทำได้โดยการเติมเอนไซม์ที่ตัด DNA ทั้งแบบจำเพาะ (ลบบางส่วน) และแบบไม่จำเพาะ (ลบทั้งหมด) และสามารถเติมข้อมูลชุดใหม่ (reload) ลงไปใน SDC ได้เช่นกันสำหรับการอ่านครั้งถัดไป กล่าวคือเราสามารถใช้ composite material อย่าง SDC-DNA ในการอ่าน ลบ (บางส่วน) เขียนใหม่ ซ้ำ ๆ ไปได้เรื่อย ๆ ซึ่งทางทีมวิจัยก็ได้ทดลองอ่านซ้ำด้วย IVT กว่าสิบครั้งก็ยังคงได้ข้อมูลที่มีคุณภาพสูงอยู่ กระบวนการที่ถูกออกแบบมาอย่างแยบยลนี้จึงช่วยให้ทีมวิจัยสามารถใช้ SDC-DNA ในการคำนวณเพื่อแก้ปัญหาในเกมระดับง่าย ๆ อย่างสุโดกุ (Sudoku) หรือหมากรุก (Chess) เพื่อเป็นการแสดงขีดความสามารถของการคำนวณด้วยระบบชีวภาพ (Biocomputing) ออกมาให้คนอ่านอย่างเราได้อ้าปากค้างกันไปพักใหญ่ ด้วยความที่การคำนวณระบบชีวภาพนี้มีเทคนิคที่ซับซ้อนระดับหนึ่ง เลยอาจจะมีคำศัพท์เทคนิคเข้ามาอธิบายมากสักหน่อยนะครับ
ในงานนี้ทีมวิจัยทดลองแก้ปัญหาในเกมกระดานขนาด 3×3 โดยใช้หลักการของหมากรุก (Chess) และสุโดกุ (Sudoku) มาใช้ โดยการแบ่งชิ้นส่วนของ DNA ออกเป็นกลุ่มที่บ่งชี้ถึงตำแหน่งบนตาราง และมีลำดับ DNA อีกส่วนหนึ่งที่บอกว่าบนตำแหน่งนั้นจะมีหมากหรือตัวเลขอะไรอยู่ ในที่นี้ก็จะมีความเป็นไปได้ 3 รูปแบบ เช่นในกรณีของ Sudoku ก็จะเป็นเลข 1, 2, หรือ 3 ส่วนหากเป็นหมากรุกก็จะเป็น knight (ม้า), bishop (บาทหลวง), หรือไม่มีหมากอยู่ (null) ทางทีมวิจัยตั้งโจทย์ออกมา 3 ข้อ โดยข้อแรกและข้อสองขึ้นกับหลักการเล่นหมากรุก ข้อแรกให้มี knight สีขาวตั้งอยู่มุมซ้ายบน ส่วนข้อสองเติม bishop สีขาวไว้ใต้ knight อีกตัวหนึ่ง แล้วตั้งคำถามว่าจะทำอย่างไรให้วางหมากสีดำโดยที่จะไม่ถูกโจมตีโดยหมากสีขาวที่ตั้งไว้อยู่แล้ว สำหรับข้อสามเป็น Sudoku กระดานง่าย ๆ คือมีเลข 4 ตัวอยู่บนกระดานเดิมแล้วให้เติมตัวเลขที่เหลือให้ตรงกับหลักการของ Sudoku หากว่าเราเคยเล่น Chess หรือ Sudoku มาบ้าง เราคงจะพอวาดคำตอบออกมาได้ไม่ยาก แต่เราจะทำอย่างไรให้ DNA ที่เราเก็บข้อมูลไว้อย่างนี้บอกชุดคำตอบที่ถูกต้องออกมาได้ล่ะ ทางทีมวิจัยเล่าว่าเขาย้อนกระบวนการไปถึงงานวิจัยที่ตีพิมพ์เมื่อปี 2000 นู้น ที่ใช้ RNase H เอนไซม์ที่จะตัดเฉพาะสายคู่ระหว่าง DNA กับ RNA เท่านั้น ประกอบกับการที่ระบบ SDC-DNA นี้สามารถใช้ IVT สร้าง RNA ได้เรื่อย ๆ ทางทีมจึงได้ติดตั้ง promoter ที่จำเพาะกับโจทย์แต่ละข้อเอาไว้ (T7, Sp6, และ T3 promoters) เวลาที่ทำ IVT จะได้อ่านถูกข้อด้วยเอนไซม์ที่จำเพาะกับ promoter ข้อนั้น โดยชุดข้อมูลนั้นมีชุดคำตอบที่เป็นไปได้ทั้งหมดติดตั้งอยู่ใน SDC-DNA อยู่เดิมแล้วจะได้ไม่ต้องกลับไปแก้ไขที่ตัวเก็บข้อมูล คล้ายกับเวลาเรามั่ว password เวลาไม่ได้เข้าไปเล่นเกมออนไลน์นาน ๆ วิธีการหาทำตอบที่ถูกก็คือการป้อนคำสั่งโดยการโยนชุด DNA สังเคราะห์ขึ้นมาลงไปในระบบเพื่อให้เกิดสายคู่ DNA-RNA duplex พร้อมกับ RNase H และทำลายชิ้นส่วน DNA-RNA ที่ไม่ใช่คำตอบสุดท้ายทิ้งไปก่อนจะนำไปอ่านข้อมูล ตรงนี้เองที่เป็นจุดที่ Biocomputing ได้ทำงาน การที่ทีมวิจัยเลือกที่จะเติม DNA เฉพาะชุดที่เป็นคำตอบที่ผิดลงไปในหลอดทดลอง ดังนั้นเจ้า enzyme RNase H จึงกำจัดสาย RNA ที่เป็นคำตอบที่ผิดออกไป เหลือเพียงคำตอบที่ถูกต้องที่จะถูกแยกออกมาเพื่อนำไปอ่านด้วย Nanopore ต่อไป ผลงานชิ้นนี้นับว่ามีหลักการล้ำ ๆ อยู่หลายจุด จัดว่าเป็นหมุดหมาย (milestone) ที่สำคัญของวงการชิ้นหนึ่งเลยทีเดียว
โรเนียวข้อมูลด้วยตัวเรียงพิมพ์ DNA ที่ใคร ๆ ก็ใช้เป็น
อีกผลงานหนึ่งที่น่าสนใจตีพิมพ์ในช่วงปลายปี 2024 จากทีมวิจัยรวมทั้งจากรัฐ Arizona ในอเมริกา จาก Beijing ประเทศจีน และจาก Stuttgart ในเยอรมนี ได้ยกกระบวนการดัดแปลง DNA ที่อยู่นอกเหนือไปจากรหัส DNA ทั่วไปอย่างกระบวนการ DNA Methylation(4) ที่ส่งผลต่อการแสดงออกของยีนและนำไปสู่การศึกษาพันธุศาสตร์แขนงใหม่ที่เรียกว่า Epigenetics (epi- เป็นคำกรีก แปลว่า เหนือ หรือ over/on ในภาษาอังกฤษ) โดยในงานวิจัยนี้ทางทีมได้เสนอกระบวนการเขียนแบบใหม่ แทนที่จะยึดกับการเขียนรหัส ATGC ที่ยังคงต้องพัฒนาไปอีกสักระยะจึงจะคุ้มค่ากับการเอามาเก็บข้อมูล ทำไมถึงไม่ลองใช้การเขียนลงไปบน DNA อย่างที่กลไกลทางธรรมชาติใช้ในกระบวนการ Epigenetics ดูล่ะ ในงานนี้ทางทีมได้ออกแบบชุด DNA ที่สามารถเติมหมู่ methyl ด้วยเอนไซม์ได้ตามการออกแบบขึ้นมา ทำให้สามารถเขียนทับลงบน DNA ได้หลาย ๆ ชุดพร้อมกัน แล้วจึงจัดกลุ่มข้อมูลของชุด DNA ในรูปแบบของ Epi-bits ก็คือถ้ามีหมู่ methyl ก็นับเป็น 1 ถ้าไม่มีก็นับเป็น 0 หากอ่านสาย DNA ยาว ๆ ก็จะเห็นรหัสคล้าย ๆ กับที่อ่านได้จากคอมพิวเตอร์ทำให้รูปแบบของการเขียนอ่านข้อมูลเป็นไปได้ไม่ซับซ้อนเหมือนกับการอ่านข้อมูล DNA ที่จะต้องมีการแปลรหัส ATCG แบบออกมาเป็นรหัส 0/1 ที่ใช้ในระบบคอมพิวเตอร์อยู่แล้ว ตรงนี้ผู้ใช้จึงสามารถลงมือแปลงและถอดรหัสโดยไม่จำเป็นต้องเรียนรู้ระบบการแปลงข้อมูลระหว่าง DNA sequence กับ Digital data
การออกแบบดังกล่าวนี้สามารถเกิดขึ้นได้ก็เพราะว่าเรามีเทคโนโลยีการอ่าน DNA methylation pattern ที่ราคาถูกลงมากอย่าง Oxford Nanopore Technology ที่นอกจากจะอ่านข้อมูลของ DNA ได้ระดับโมเลกุลเดี่ยว (single molecule) ในสารละลายที่ปนกันแล้ว ยังช่วยบอกได้ด้วยว่าลำดับ DNA ส่วนไหนมีการเติมหมู่ methyl ลงไปโดยที่ไม่ต้องเพิ่มสารเคมีพิเศษลงไปเลย(5) เพราะว่าการอ่านข้อมูลแบบ ONT นี้จะให้ข้อมูลออกมาในรูปแบบของกระแสไฟฟ้าที่เปลี่ยนแปลงไปการผ่านช่องแคบของสาย DNA นั่นเอง ลองนึกถึงการพายเรือข้ามคลองเล็ก ๆ ถ้าเรือรูปร่างต่างกันก็จะส่งผลต่อกระแสน้ำต่างกันออกไปคล้ายกับกระแสไฟฟ้าที่ได้ผลกระทบจากลักษณะของ DNA ดังนั้นการเพิ่มหมู่ methyl ลงไปจึงทำให้โครงสร้างทางเคมีของ DNA ใหญ่ขึ้นและส่งผลกับคลื่นกระแสไฟฟ้าของแต่ละชิ้นส่วน DNA ที่เรานำมาวิเคราะห์ด้วย ONT ด้วยทางทีมวิจัยมุ่งหวังจะให้กระบวนการนี้เก็บข้อมูลได้มาก ๆ จึงทำการประยุกต์ใช้หลักของการเรียงพิมพ์ (typesetting) ที่แพร่หลายในการพิมพ์ในยุคตั้งต้นมาใช้ ในขั้นต้นนั้น ทางทีมได้สร้าง DNA ชิ้นเล็ก ๆ เหมือนกับการสร้างแป้นพิมพ์แล้วนำชิ้นส่วนเหล่านี้มาเรียงต่อกันบน DNA อีกเส้นหนึ่งเหมือนกับที่โรงพิมพ์สมัยก่อนต้องเอาตัวอักษรมาเรียงไว้บนแป้นพิมพ์ จากนั้นจึงนำเอากระดาษ (DNA อีกเส้น) มาคัดลอกหมึกด้วยเอนไซม์ (methylation pattern) ลงไปตามลำดับอักษรที่เรียกไว้ เมื่อนำเอา algorithm อื่น ๆ ที่ใช้ในวงการเก็บข้อมูล DNA อย่างการติดบาร์โค้ด (DNA barcoding) ที่ช่วยเหมือนกับการเขียนตัวเลขไว้บนเซียมซี DNA แต่ละเส้น ทำให้เราสามารถเอาข้อมูลที่มีมาเรียงเป็นรูปร่างขึ้นมาได้ และหากทำการเรียงพิมพ์นี้พร้อม ๆ กันหลายร้อยหลายพันชิ้นก็จะสามารถป้อนข้อมูลลงบนชุดเซียมซีที่ใหญ่ขึ้นได้จนได้รูปที่มีความละเอียดชัดขึ้นไปอีก สุดท้ายทีมวิจัยก็ยังนำเอากระบวนการที่ว่าไปให้อาสาสมัครทดลองออกแบบและลงมือพิมพ์ข้อมูล Epi-bits ลงบนเส้น DNA ขึ้นมาจริง ๆ แม้ว่าการอ่านผลจะพบว่ามีการพิมพ์ผิดอยู่บ้างแต่ก็สามารถแก้ไขสะกดผิดในประโยคเหล่านั้นด้วย Large Language Model AI ได้อย่างง่ายดาย ผลงานชิ้นนี้นับว่าเป็นการประยุกต์ทั้งศาสตร์ทางชีววิทยาสังเคราะห์และศิลปะของการพิมพ์ที่มีการพัฒนามาหลายร้อยปีมามัดรวมกันในการเก็บข้อมูลลงบน DNA ได้อย่างมีสีสันจนหลายคนแห่กันแชร์รูปหมีแพนด้าในงานจนไวรัลกันไปพักหนึ่งเลยทีเดียว
ก้าวข้ามขีดจำกัดด้วยการเพิ่มรหัส DNA แล้วเอาไปเก็บไว้ในสิ่งมีชีวิต
งานทั้งสองชิ้นนี้ที่ยกมานี้เป็นตัวอย่างการออกแบบกระบวนการเก็บข้อมูลบน DNA ที่แฝงหลักการผสมผสานระหว่างองค์ความรู้หลายแขนงเข้าด้วยกัน และยังได้รับแรงผลักดันจากเทคโนโลยีใหม่ (enabling technology) อย่าง ONT เข้ามาช่วยให้การออกแบบนี้เรียบง่ายและคุ้มค่าใกล้กับการใช้งานจริงมากขึ้น นอกเหนือจากด้านการจัดเก็บข้อมูลแล้ว การพัฒนาวงการ DNA Data Storage ยังสามารถทำได้ในอีกหลายแง่มุมทั้งด้านการขยายขีดจำกัดของรูปแบบ DNA ไปจนถึงกระบวนการจัดเก็บในสิ่งมีชีวิต ตัวอย่างที่สำคัญของการออกจากกรอบธรรมชาติคือการคิดค้นรหัส DNA ชุดใหม่จาก 4 อักขระให้เป็น 8 อักขระ (DNA Hachimoji) ในปี 2019(6) ที่สามารถถอดรหัสจาก DNA ให้เป็น RNA ได้ จนไปถึงการพัฒนาล่าสุดเพิ่มเป็น 12 อักขระที่ใช้เอนไซม์เขียนพร้อมกับการคิดค้นวิธีอ่านด้วย ONT ไปพร้อม ๆ กัน(7) ในด้านของการเก็บของมูลในสารพันธุกรรมของสิ่งมีชีวิตเองก็เป็นอีกกระบวนการหนึ่งที่น่าสนใจ ด้วยกระบวนการเพิ่มจำนวนและซ่อมแซม DNA นั้นมีความจำเป็นในการแบ่งเซลล์และเจริญเติบโตของสิ่งมีชีวิตทุกชนิด การเก็บและคัดลอกข้อมูลในเซลล์จึงมีความน่าสนใจไม่แพ้การเก็บ DNA ในหลอดทดลอง การบันทึกข้อมูลในเซลล์นี้สามารถเขียนลงบนจีโนม (Genome) โดยตรง หรือโดยการเขียนลงบนพลาสมิด (Plasmid) ที่สามารถแยกชิ้นส่วนออกมาจากจีโนมมาวิเคราะห์ด้วยเทคโนโลยี ONT ได้ในราคาที่ถูกลงเรื่อย ๆ บริษัท Plasmidsaurus ที่ถูกกล่าวถึงในช่วงแรก ๆ ของบนความนั้นให้บริการอ่าน plasmid ทั้งวงในราคาเพียงแค่ 15$ ต่อหนึ่งหลอด ซึ่งในหลอดนั้นอาจจะใส่ Plasmid DNA ไปมากกว่าหนึ่งชิ้นก็ได้ การบรรจุ Plasmid DNA ลงในแบคทีเรียนั้นมีหลักการที่ต้องคำนึงถึงอยู่หลายประเด็นซึ่งอาจจะนำมาเล่าให้ฟังในวาระถัดไป(8) (หากบทความที่ผมส่งไปได้รับตีพิมพ์นะครับ)
ในยุคสมัยที่ผันเปลี่ยนไปอย่างรวดเร็วตั้งแต่ที่เราเก็บข้อมูลได้แค่หลัก Megabytes ใน Floppy Disk (ที่มาของไอคอน Save ในโปรแกรมต่าง ๆ ใครเกิดไม่ทันลองไปอ่านดูนะ) มาจนยุคที่เราเก็บใน Thumb Drive ที่ขนาดเท่านิ้วมือแต่เก็บข้อมูลได้หลัก Terabytes การเก็บข้อมูลยุคอนาคตอันใกล้อาจจะเปลี่ยนไปเป็นหลอด DNA ที่เก็บข้อมูลระดับปโกฎิไบต์เอาไว้ห้อยคอแทนพระเครื่องก็เป็นได้ ส่วนตัวผมเองก็คงเก็บ DNA แคปซูลที่บรรจุหนังสือเล่มแรกที่เขียนด้วย DNA เอาไว้เป็นจดหมายถึงตัวเองในอนาคต เผื่อว่าสักวันหนึ่งจะทุบมันออกมารำลึกความหลังสมัยที่เรายังไม่มีเครื่องพิมพ์อักขระชีวภาพ (DNA Printer) ไว้ใช้ทั่วไป คล้ายกับการที่เราสามารถมีเครื่องพิมพ์กระดาษแทบทั่วทุกบ้านในทุกวันนี้ ถึงแม้ว่าก่อนการเรียงพิมพ์ (Typesetting) จะถูกคิดค้นโดย Johannes Gutenberg ในศตวรรษที่ 15 เรายังต้องคัดลอกเอกสารด้วยมือแบบที่สมัยเด็ก ๆ เราต้องลอกการบ้านจากเพื่อนลงสมุดส่งครูกันทุกเช้า แฮร่!!! ผมหมายถึงลอกโจทย์ครับ คำตอบเขียนเอง

References
(2) Morgan, S. The World Will Store 200 Zettabytes Of Data By 2025. Cybercrime Magazine.
(8) Kiattisewee, C. How Many Plasmids Can Bacteria Carry? A Synthetic Biology Perspective. Manuscript Submitted.